Wenn es darum geht, Künstliche Intelligenz erklärbar zu machen, liegt der Fokus häufig darauf, den Grund für die von einem Modell getroffene Entscheidung herauszufinden (siehe hierfür auch unseren Blogbeitrag „Warum KI erklärbar sein muss“). Gehen wir einen Schritt zurück, also zur Erstellung eines solchen Modells, befinden wir uns beim sogenannten Modell Training. Um nachvollziehen zu können, wie ein Modell entstanden ist, sollten die Metadaten eines Trainingsvorgangs festgehalten werden. Hierbei spricht man auch von Experiment Tracking. Für einen Trainingsvorgang können Metadaten, wie verschiedene Parameter, Hyperparameter und Metriken getrackt werden. Liegen genügend Informationen vor, kann mit einem wiederholten Trainingsvorgang das gleiche oder zumindest ein vergleichbares Ergebnis erzielt werden. Dieser Umstand ist auch für die Evaluation von Modellen wichtig, die durch verschiedene Traingingsvorgänge entstanden sind. Unterschiede in den Ergebnissen können so besser auf die voneinander verschiedenen Daten oder Metadaten zurückgeführt werden. Durch eine klare Übersicht über die Ergebnisse des Modells, kann das beste Modell ausgewählt werden.
Häufig stehen in der explorativen Phase des ML-Engineering-Prozesses das Modelltraining und das Erzielen guter Metriken im Vordergrund, während die Dokumentation der Ergebnisse in den Hintergrund fällt. Eine gute Dokumentation zeichnet sich jedoch besonders bei langwierigen und komplexen Prozessen aus und hilft dabei, den Überblick zu bewahren und somit am Ende ein nachvollziehbares Ergebnis zu erzielen.
Zurzeit erfolgt die Dokumentation von Experimenten in vielen Fällen noch in Form von Textdateien und verschiedene Arbeitsstände werden häufig durch Suffixe an den Dateinamen, wie zum Beispiel “Modell_v1, Modell_v2, Modell_v2_approach1, …” markiert.
Eine automatische Aufzeichnung der Trainingsvorgänge und das Versionieren von Arbeitsständen können die Arbeit erleichtern und führen zu einer übersichtlichen Dokumentation. Ein Tool, das bei diesen Schritten unterstützen kann, ist MLflow. Dieses Werkzeug wollen wir euch anhand eines Beispielworkflows in diesem Beitrag vorstellen.
Der veranschaulichende Beispielworkflow kann anhand von Codebeispielen auf dem eigenen Rechner nachvollzogen werden. Hierfür wird eine Linux-Umgebung empfohlen. Zudem sollten Git und Anaconda bereits installiert sein.
Ein Beispielworkflow in MLflow
Ziel dieses Beispielworkflows soll es sein, mithilfe des Wine-Quality-Datensatzes ein Modell für die Einordnung von Weinsorten in Qualitätsstufen zu entwickeln. Hierbei handelt es sich um ein Klassifikationsproblem (siehe „Welche Arten vom Maschinellen Lernen gibt es?“ für mehr Informationen über Klassifikation.). Den Weinen wird anhand der Eingabevariablen fixed acidity (feste Säuren), volatile acidity (flüchtige Säuren), citric acid (Zitronensäure), residual sugar (Restzucker), chlorides (Chloride), free sulfur dioxide (freies Schwefeldioxid), total sulfur dioxide (Gesamtschwefeldioxid), density (Dichte), pH (pH-Wert), sulphates, alcohol (Alkoholgehalt) die Ausgabevariable quality (Qualitätsnote) mit einem Wert zwischen 0 und 10 zugeordnet. Als Grundlage nutzen wir den Code aus dem offiziellen MLflow Repository.
SCHRITT 1: Einrichten des Codes und MLflow
Zuerst gilt es, den Code und MLflow einzurichten. Den Code erhalten wir, indem wir die zentrale Codeablage von MLflow beziehen. Das sogenannte Repository können wir über eine beliebige Kommandozeile mit Git klonen. Dazu sollten wir uns jedoch zuerst in den Ordner fortbewegen, in welchem wir den Code ablegen möchten. Nach dem Klonen bewegen wir uns dann in das Verzeichnis „./mlflow/examples/sklearn_elasticnet_wine/.“ Hierin befinden sich bereits eine Notebook-Datei train.ipynb und eine conda.yaml-Datei, mit der wir eine vordefinierte Anaconda-Umgebung namens tutorial erstellen können. Das machen wir mit dem Befehl conda env create -f conda.yaml.
Um eine erstellte Conda-Umgebung im Jupyter-Notebook ansprechen zu können, muss zunächst der Ipython-Kernel in unserer Umgebung installiert werden; anschließend wird für diesen die aktuelle Umgebung unter einem Namen, z.B. ebenfalls tutorial, installiert und somit im Jupyter-Notebook ansprechbar gemacht. Wenn noch nicht getan, kann dann zusätzlich jupyterlab zum Aufrufen der Jupyter-Notebooks installiert werden.
Anschließend starten wir unser Jupyter-Notebook mit dem Befehl jupyter notebook.
Das Jupyter-Notebook läuft jetzt im aktuellen Kommandofenster, in dem auch eine URL bereitgestellt wird, über welche die Nutzeroberfläche des Jupyter-Notebooks im Browser aufgerufen werden kann.
In einem neuen Kommandofenster bewegen wir uns nun erneut in den Ordner sklearn_elasticnet_wine. Damit die von uns gewünschten Informationen getrackt werden, sollten wir die tutorial-Umgebung aktivieren, da MLflow in dieser bereits installiert ist. Aus dem Ordner sklearn_elasticnet_wine können wir dann die MLflow-Nutzeroberfläche mit dem Befehl mlflow ui aufrufbar machen. Hiermit wird der sogenannte Tracking-Server gestartet, der dann standardmäßig über http://localhost:5000 erreichbar ist. MLflow legt die Informationen zu den getrackten Trainingsvorgängen im Ordner mlruns ab. Diese werden von MLflow geladen und auf dem Tracking-Server dargestellt.
Unter Windows funktionieren die aufgeführten Kommandos oft nicht problemlos. Möglicherweise helfen folgende Punkte bei der Ausführung:
- Den Pfad zur Anacondainstallation setzen, wenn Anaconda angenommen im Ordner C:tools installiert wurde: set PATH=%PATH%;C:toolsAnaconda3;C:toolsAnaconda3Scripts
- Die Conda-Umgebung „tutorial“ ohne expliziten „conda“-Aufruf aktivieren: activate tutorial
- Folgende Dateien aus C:toolsAnaconda3Librarybin in C:toolsAnaconda3DLLs kopieren:
- libcrypto-1_1-x64.*
- libssl-1_1-x64.*
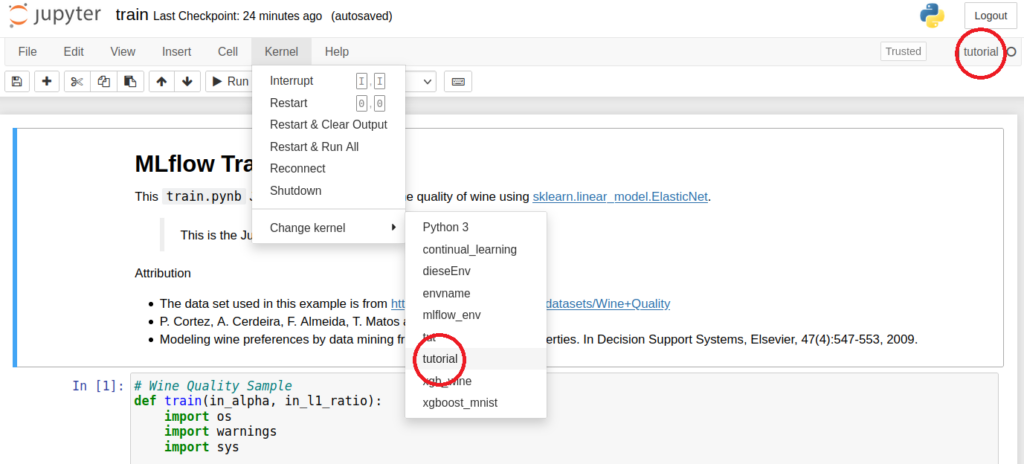
Jupyter Notebook „train“ mit aktiviertem „tutorial“-Kernel
SCHRITT 2: Ein Jupyter Notebook für die Nutzung mit MLflow einrichten
Damit wir das Notebook uneingeschränkt nutzen können, muss unsere Conda-Umgebung im Notebook aktiviert werden. Dies haben wir bereits im ersten Codeblock ermöglicht. Nun können wir die Nutzeroberfläche des Jupyter-Notebook mit der bereitgestellten URL aus Schritt 1 öffnen und über „Kernel“ à „Change kernel“ unseren „tutorial“-Kernel wählen und das Notebook für das Tracking vorbereiten. Die zu trackenden Informationen müssen wir im Notebook definieren. Betrachten wir das train.ipynb-Notebook, erkennen wir, dass wir zuerst die Funktion mlflow.start_run() aufrufen. Diese startet einen Run, für den der anschließende eingerückte Code ausgeführt wird. In unserem Fall umfasst der Run einen Trainingsvorgang. Bei einem Fehler oder nach Ausführen des eingerückten Codes wird der Run automatisch beendet. Der eingerückte Code ruft die Funktionen mlflow.log_param() sowie mlflow.log_metric() auf, um Parameter und Metriken zu loggen, also aufzuzeichnen, und abschließend mlflow.sklearn.log_model() um das spezifische sklearn-Modell zu sichern.
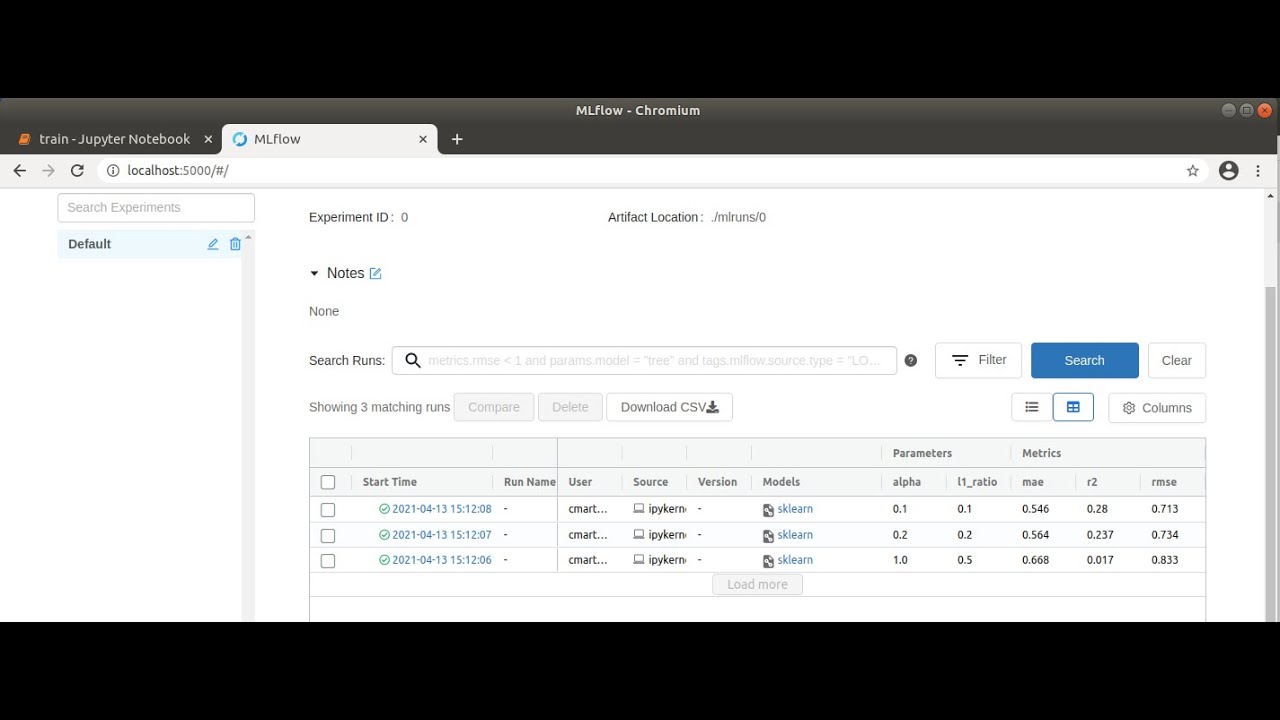
In den folgenden Zellen des Notebooks werden drei Trainingsvorgänge mit verschiedenen Werten für die Parameter alpha (Höhe der Strafe für Einbeziehung irrelevanter Merkmale) und l1_ratio (Gewichtung zwischen den zwei standard Regularisierungsmethoden L1 und L2) definiert. Führen wir das gesamte Notebook aus, werden diese Trainingsvorgänge als Runs in MLflow abgelegt, die wir in der MLflow-Nutzeroberfläche betrachten können.
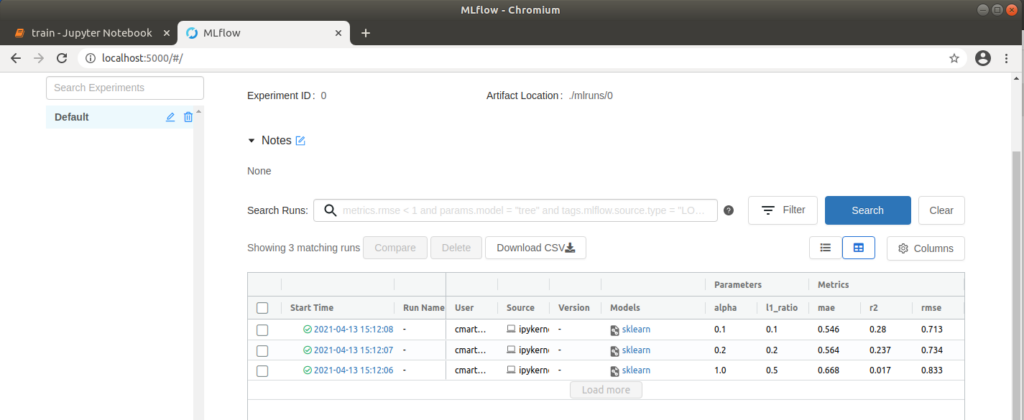
SCHRITT 3: Ergebnisse in der MLflow Nutzeroberfläche betrachten
Nun können wir in unserem Browser http://localhost:5000 aufrufen und in der MLflow-Oberfläche das Projekt “Default” auswählen, um die ausgeführten Runs zu vergleichen. Dazu warten wir zunächst, bis an allen Runs ein grüner Haken erscheint, der signalisiert, dass die Vorgänge abgeschlossen sind. Danach wählen wir alle drei Runs aus und klicken auf “Compare”. Im sich öffnenden Fenster werden Parameter, die sich in den drei Runs unterscheiden, farblich hervorgehoben. Das sind in unserem Fall die beiden Parameter alpha und l1_ratio. Über verschiedene Plots können wir die Runs auch visuell vergleichen. Klicken wir beispielsweise auf „rmse” (Wurzel [root] des mean squared error), wird eine Grafik angezeigt, auf der klar zu erkennen ist, dass der „rmse” für Run 1 am niedrigsten ist. Das bedeutet, dass die Differenz zwischen beobachteten und vorhergesagten Daten am geringsten und somit das Modell am besten auf die verwendeten Daten angepasst ist. Auf der linken Seite lassen sich noch weitere Einstellungen machen; so können beispielsweise zusätzlich noch „r2” (Bestimmtheitsmaß), ebenfalls zur Beurteilung der Anpassung des Modells auf die Daten, und „mae“ (mean absolute error) für die Beurteilung der Genauigkeit der Vorhersagen des Modells, angezeigt werden. Je nach Anwendungsfall bietet es sich an, verschiedene Darstellungsmöglichkeiten auszuprobieren.
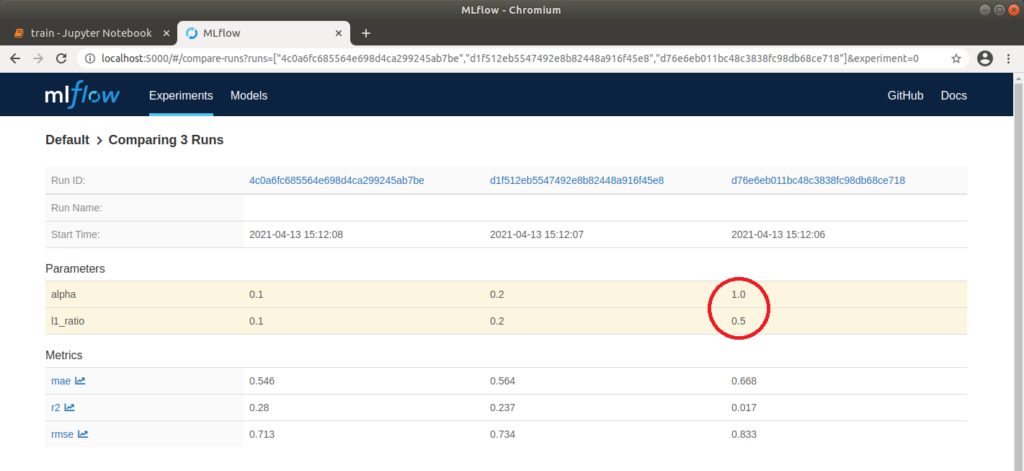
MLflow: Tabellarischer Vergleich der Parameter und Metriken dreier Runs
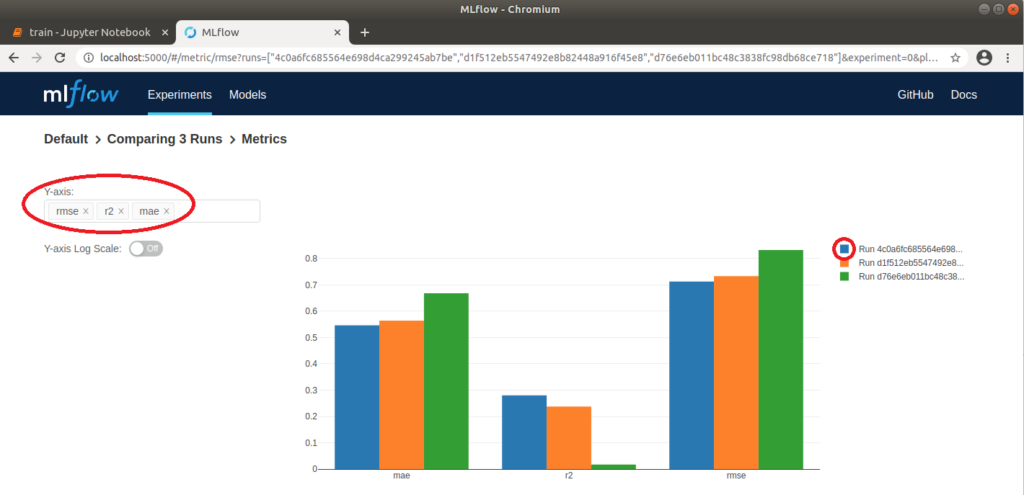
SCHRITT 4: Besten Run detailliert betrachten
Der Run 1 mit den Werten alpha=0.1 und l1_ratio=0.1 hat mit einem „rmse” von ca. 0.71 das beste Ergebnis geliefert. Über die Run ID können wir Metainformationen zu diesem Run explizit aufrufen. Klicken wir auf die oben genannte Grafik, öffnet sich außerdem ein Pop-Up, über das wir auch direkt zu einem Run springen können. Hier können wir Notizen hinzufügen und die genauen Parameter und Metriken erneut betrachten. Unter dem Punkt „Artifacts“ können wir außerdem die entsprechenden Modelldateien betrachten. Auch eine conda.yaml kann mitgeliefert werden, unter der das Modell auf anderen Rechnern geladen oder trainiert werden kann. Diese wird ohne explizite Definition automatisch erzeugt. Mit einem Klick auf den Ordner „model” wird außerdem ein Python-Codebeispiel angezeigt, durch das eine Vorhersage erfolgen kann.
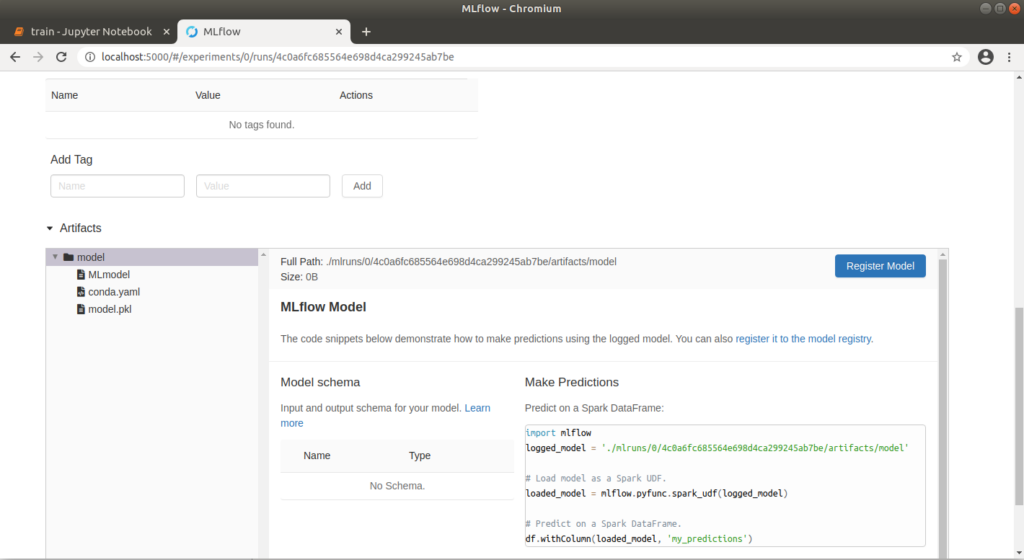
In MLflow abrufbares Modell-Artefakt
SCHRITT 5: Ein Modell von MLflow beziehen und für die Vorhersage nutzen
Um das von uns erstellte Modell zu beziehen und damit eine Vorhersage zu machen, können wir uns am Codevorschlag von MLflow orientieren. Wir passen diesen an, indem wir die passende run_id aus der URL beziehen (http://localhost:5000/#/experiments/experiment_nummer/runs/run_id) und im Code einfügen. Den Code schreiben wir in eine neue Zelle in unserem Notebook. Die Eingabedaten für das Modell erzeugen wir im Code manuell als input_data:
Nach dem Ausführen der Zelle erhalten wir die Bewertung für den von uns definierten Wein. Spielen wir ein wenig herum, merken wir schnell, dass das Modell schon allein durch einen sehr hohen oder niedrigen Alkoholgehalt in seiner Bewertung beeinflusst wird. 🙂
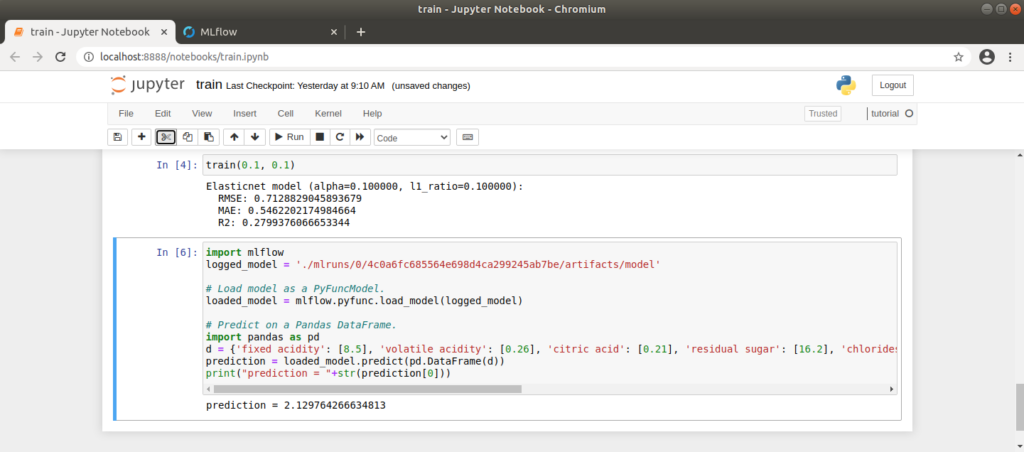
Für die manuell angelegten Eingabedaten liefert das bezogene Modell hier einen Wert von ca. 2.1
Tracking, Tracking, Tracking, …
Unsere Experimente können mithilfe weniger zusätzlicher Codezeilen automatisch durch MLflow getrackt werden. Dadurch haben wir einen besseren Überblick darüber, welche Metriken wir mit welchen Parametern erreichen, und wir können unsere entwickelten Modelle einem konkreten Trainingsvorgang zuordnen, dem wir wiederum eine konkrete Trainingsumgebung zuordnen können.
Abschließend ist zu erwähnen, dass hier mit dem statischen Wine-Quality-Datensatz gearbeitet wurde. Genauso wie sich Modelle und Parameter in der realen Welt verändern können, ist jedoch auch eine Veränderung des Sourcecodes und der zugrundeliegenden Daten möglich. In den nächsten Blogeinträgen dieser Reihe, gehe ich deshalb darauf ein, wie Source Code und Daten während der Exploration verwaltet werden können.
Mehr Informationen zur Implementierung im zugehörigen Video: