„Time is brain“ – dieser bekannte Satz fasst die Problematik eines Schlaganfalls zusammen. Erleidet eine Person einen Schlaganfall, kommt es auf schnelles Handeln an, um bleibende Schäden wenigstens zu vermindern. Ursache für einen Schlaganfall kann entweder ein Gefäßverschluss (ischämisch) oder eine Blutung im Gehirn (hämorrhagisch) sein. Bei einem Gefäßverschluss kann die Durchblutung durch blutverdünnende Medikamente wiederhergestellt werden. Dieses Medikament wäre im Falle einer Blutung jedoch kontraproduktiv. Deswegen muss eine bildgebende Diagnostik angefertigt werden, um möglichst schnell zwischen den beiden Krankheitsbildern zu unterscheiden und bei einem hämorrhagischen Schlaganfall die Lokalität und Größe der Blutung untersuchen zu können. Dazu wird in der Regel eine Computertomografie (CT) (s. Abb. 1) des Schädels erstellt. Diese besteht aus vielen einzelnen Bildern (Schichten), die zusammen den dreidimensionalen Eindruck des Kopfes erzeugen.
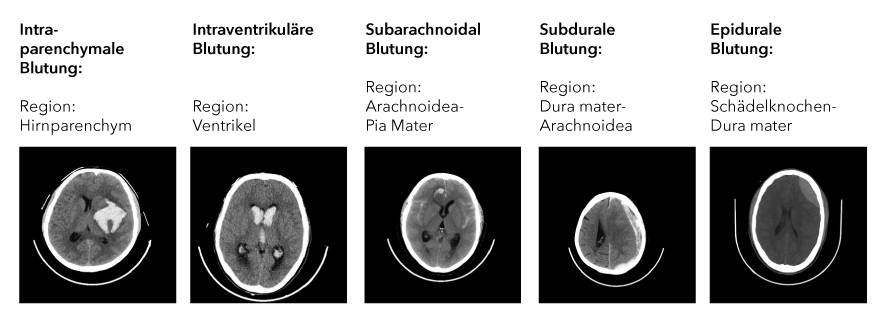
Abbildung 1: CT Aufnahmen des Schädels mit Blutungen in unterschiedlichen Regionen des Kopfes.
Die Analyse des CTs kann gerade für ärztliche Berufseinsteigende zeitaufwändig und herausfordernd sein. Aus dieser Problematik entstand die Idee, Ärzt*innen durch eine Anwendung zu unterstützen, welche automatisch klassifiziert, ob auf einer Computertomografie eine Blutung vorliegt. Dazu bietet sich der Einsatz von Deep Learning und insbesondere von Convolutional Neural Networks (CNNs) an. In einem sicherheitskritischen Bereich wie der Medizin muss gleichzeitig sichergestellt werden, dass Ärztinnen die Vorhersagen eines solchen Systems nachvollziehen können.
Woher kommen die Daten und wie funktioniert das Modell?
Als Grundlage für die Realisierung einer solchen Anwendung werden zunächst Daten benötigt. Die Radiological Society North America (RSNA) veröffentlichte im Jahr 2018 im Rahmen des Wettbewerbs „RSNA Intracranial Hemorrhage Detection“ auf der Plattform Kaggel den größten multinationalen und multi-institutionellen Datensatz von CT-Scans mit mehr als 25.312 CTs, die aus insgesamt 874.035 Bildern bestehen. Diese Bilder wurden von Radiolog*innen mit sechs verschiedenen Klassen gekennzeichnet. Die erste Klasse gibt an, ob allgemein ein Schlaganfall vorliegt und die anderen Klassen jeweils, in welcher Region des Kopfes die Blutung auftritt (s. Abb. 1).
Neben dem Datensatz wird als Grundlage für das Modell zur Klassifizierung von hämorrhagischen Schlaganfällen der Siegeralgorithmus dieses Wettbewerbs verwendet. Weitere Informationen zum Modell finden sich auch im Paper von Wang et al., welches zum Zeitpunkt dieser Arbeit noch nicht veröffentlicht war. Das im Rahmen des Wettbewerbs veröffentlichte Modell wird neu trainiert, indem der ursprüngliche Trainingsdatensatz in je einen neuen Trainings- und einen Testdatensatz aufgeteilt wird. Die grundlegende Struktur des Modells ist in Abb. 2 dargestellt. Zunächst werden aus den medizinischen Daten (DICOM-Format) einerseits die Bilddaten, anderseits Metadaten extrahiert. Zu den Metadaten gehört hier beispielsweise die anonyme Patienten-ID. Im nächsten Schritt werden die Bilddaten vorverarbeitet. Die vorverarbeiteten Bilder sowie die Metadaten dienen als Eingabe für ein CNN-Modell. Das CNN-Modell verarbeitet alle Schichten des CTs unabhängig voneinander und erzeugt für jede Schicht eine Ausgabe, ob eine Blutung vorliegt und in welcher Region des Gehirns sie auftritt. Diese Ausgabe sowie die gelernten Merkmale des CNN-Modells sind Eingabe für ein sequenzielles Modell. Im sequenziellen Modell wird das gesamte CT mit allen Schichten als Sequenz betrachtet. Die Idee ist, die Nachbarschaftsinformationen zu nutzen, wenn zum Beispiel auf aufeinanderfolgenden Schichten des CTs die gleiche Blutungsklasse erkannt wurde und so das Modell zu verbessern. Im sequenziellen Modell können zudem die Vorhersagen und Merkmale von verschieden trainierten CNN-Modellen zusammengeführt werden. Ausgabe des sequenziellen Modells ist erneut eine Klassifikation für jede Schicht des CTs.
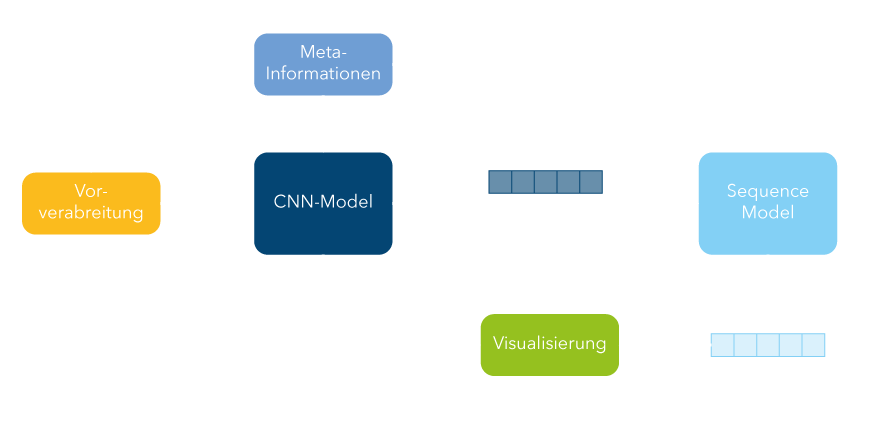
Abbildung 2: Überblick über die Struktur des Modells.
Das Modell wird mit verschiedenen Vorverarbeitungsmethoden, CNN-Architekturen und Kombinationen neu trainiert. Die Einbettung des Modells wird angepasst, sodass beliebige CTs als Eingabedaten verarbeiten werden können. Durch die Experimente ergibt sich als bestes Modell eine Kombination aus zwei verschiedenen CNN-Modellen, deren Ausgabe im sequenziellen Modell zusammengeführt wird. Beide CNN-Modelle haben als Backbone eine SE-RexNext-101-Architektur. Bei dieser CNN-Architektur wird eine ResNeXt-101-Architektur mit einem sogenannten Squeeze- and Excitation-Block (SE-Block), kombiniert, wobei wichtigere Feature-Maps stärker gewichtet werden. Im ersten Modell werden die medizinischen Eingabebilder in drei verschiedenen Fensterungen erzeugt. Alle drei heben unterschiedliche Strukturen wie z.B. Knochen oder Weichgewebe hervor, indem aus den digitalen Werten des CTs, die in der sogenannten Houndsfield-Skala vorliegen, ein spezifisches Intervall ausgewählt wird. Die Houndsfield-Skala stellt die Abschwächung von Röntgenstrahlung im Gewebe dar. Im zweiten Modell werden drei aufeinanderfolgende Schichten eines CTs gleichzeitig als Eingabe für das CNN-Modell genutzt.
Nach dem Training wird das Modell auf zwei weiteren Datensätzen evaluiert. Als erster Datensatz wird der öffentlich zugängliche CQ500 -Datensatz verwendet. Das Modell erzeugt hier ähnlich gute Ergebnisse wie für den RSNA-Testdatensatz. Über die meisten Klassen hinweg sind die Performanz-Metriken Accuracy und AUC hoch (CQ500-Datenset: Accuracy: 97-99%, AUC: 98-99%). Der F1-Score ist für fast alle Klassen hoch (71%-98%), außer für epidurale Blutungen (25%). Gleiches wird auch auf dem RSNA-Testdatensatz beobachtet. Das Modell scheint hier schlecht zu performen, da sehr viel weniger Daten für Blutungen in dieser Region vorhanden sind. Viele epidurale Blutungen werden falsch als subdural erkannt. Subdurale Blutungen und epidurale Blutungen treten nah beieinander an jeweils unterschiedlichen Seiten der äußeren Hirnhaut auf.
Als zweiter Testdatensatz werden Daten eines lokalen Krankenhauses verwendet. Um gezielt Daten aus dem normalen Klinikalltag zu betrachten, wurden über zwei Monate hinweg alle intrakraniellen CT-Bilder gesammelt. Alle CT-Bilder werden sowohl durch das neu trainierte Modell klassifiziert als auch durch einen Radiologen des lokalen Krankenhauses beurteilt. Anschließend werden beide Ergebnisse gemeinsam mit dem Radiologen analysiert.
Wie können Ärzt*innen die Entscheidungen des Modells nachvollziehen?
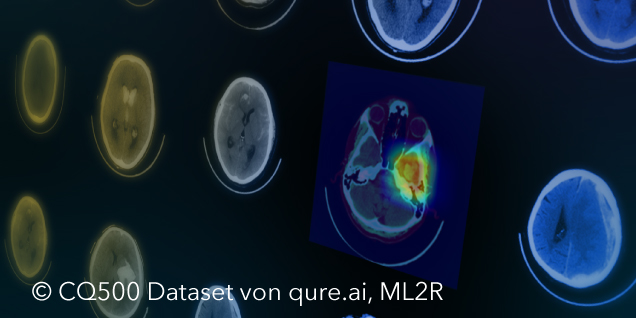
Auch wenn das Modell sehr gute Ergebnisse liefert, muss für die Diskussion mit dem Radiologen sowie für den realen Anwendungskontext die Frage geklärt werden, wie Ärzt*innen die Entscheidung des Modells nachvollziehen können. Mithilfe verschiedener Post-Hoc-Verfahren können die Entscheidungen eines Algorithmus im Nachhinein lokal erklärt werden. Dazu wird im Rahmen dieser Arbeit das Verfahren Grad-CAM++ genutzt. Hierbei wird eine klassenspezifische Visualisierung (s. Abb. 3) erzeugt, die die Wichtigkeit einer bestimmten Region für die Entscheidung des Algorithmus veranschaulicht. Die Visualisierung ist eine gewichtete Linearkombination der positiven Gradienten der Feature-Maps des letzten Convolutional Layers.
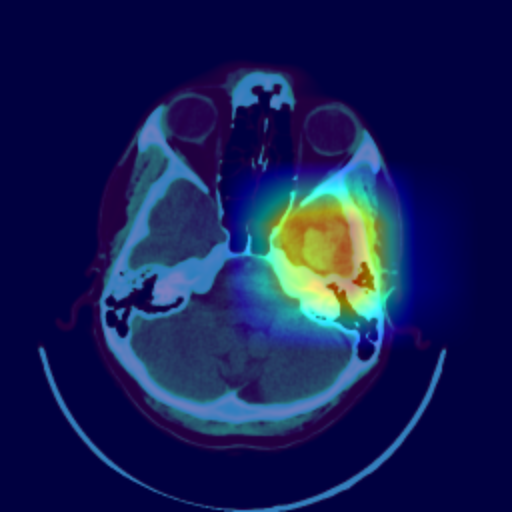
Die Visualisierung erlaubt es dem Radiologen nachzuvollziehen, welche Regionen für den Algorithmus auffällig waren und die Klassifikation zu überprüfen. In der gemeinsamen Analyse des zweiten Datensatzes kann der Radiologe beispielsweise einige vom Algorithmus detektierte Stellen als Blutung bestätigen, die im Voraus übersehen wurden. Gleichzeitig zeigt die Visualisierung, dass das Modell fälschlicherweise mehrere OP-Defekte als Blutung klassifiziert hat.
Fazit
Deep Learning liefert oftmals sehr gute Ergebnisse bei der Klassifizierung medizinischer Bilddaten. Die automatische Klassifizierung von hämorrhagischen Schlaganfällen lässt sich durch CNNs in Verbindung mit einem sequenziellen Modell realisieren und liefert in den meisten Fällen eine hohe Performanz. Für den Einsatz in einem sicherheitskritischen Bereich wie der Medizin ist neben einer durchgehend hohen Performanz allerdings auch eine Erklärung der Ergebnisse nötig. Die Visualisierung durch Grad-CAM++ bietet eine Möglichkeit, die Ergebnisse für Ärzt*innen nachvollziehbar zu machen. Es scheint sinnvoll, Medizinerinnen durch eine solche Anwendung zu unterstützen und durch eine schnelle Klassifizierung von CT-Bildern Auffälligkeiten möglichst schnell zu detektieren. Dennoch müsste medizinisches Fachpersonal die Ergebnisse in jedem Fall kritisch prüfen und bestätigen, um falsche Vorhersagen auszuschließen.
